首个JMJ家族组蛋白H3K4去甲基化酶的晶体结构解析
真核生物的基因组DNA与组蛋白结合形成染色质的基本结构,调控基因表达,行使基因的生物学功能。位于组蛋白末端的一些氨基酸会被不同的化学基团修饰,导致染色质结构的改变,影响基因表达和基因组功能,这是一种表观调控机制;而组蛋白甲基化是组蛋白修饰的一种重要要形式。因此弄清组蛋白甲基化酶和去甲基化酶如何特异地识别这些不同的赖氨酸位点是解析表观遗传机理的焦点之一。此前已有文献报道了JMJ家族H3K9、H3K27及H3K36去甲基化酶的晶体结构,并阐述了其底物特异性的分子机理,但H3K4去甲基化酶却一直没有结构信息。华中农业大学张启发课题组、周道绣课题组与清华大学娄智勇课题组合作,鉴定了水稻中的一个H3K4去甲基化酶JMJ703并解析其晶体结构。相关的研究成果于2013年1月24日在线发表于《Plos Genetics》上。
该研究通过生化分析发现,水稻JMJ703蛋白能够特异的去除组蛋白H3第4位赖氨酸位点(表示为H3K4)的甲基化。为了了解JMJ703如何行使其去甲基化功能,他们进一步解析了JMJ703核心区域(以下简称c-JMJ703)单独的晶体结构以及与辅基类似物NOG、底物H3K4me3短肽的复合物晶体结构。
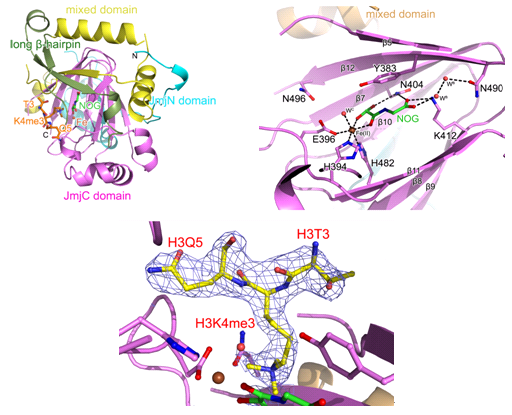
利用北京同步辐射装置获得了2.8 Å分辨率的 c-JMJ703 蛋白晶体结构。从整体上看,c-JMJ703的结构与已知结构的其他JMJD2A亚家族蛋白类似,由JMJN、long-beta sheet、mixed domain和JMJC等结构域组成。催化活性中心位于JMJC结构域中,而其余三个结构域则分布在JMJC结构域两侧,整体呈三明治状。
通过解析复合物结构,该研究鉴定出了该蛋白催化结构域中的一些关键氨基酸,它们分别参与了与辅基的相互作用以及H3K4me3短肽的特异性识别。c-JMJ703与辅基类似物NOG的复合体结构显示,该蛋白沿袭了JMJ家族组蛋白去甲基化酶中较为保守的辅基结合方式。而c-JMJ703与H3K4me3短肽的复合体结构初步揭示了该类蛋白识别H3K4me3的分子机理。通过与特异去除H3K9和H3K36甲基化的JMJ家族蛋白的复合物结构进行比较,该研究发现,c-JMJ703采用了较为不同的底物结合方式。由于JMJ703的其他结构域也可能参与底物结合,因此在c-JMJ703与H3K4me3短肽的复合体结构中,短肽的多数残基由于占有率低而不可见。
这是首次报道的H3K4位点去甲基化酶蛋白的分子结构,也是水稻中的首个表观修饰因子的晶体结构,对于表观遗传学的机理研究有着重要的意义。我们期待更多的结构信息来对H3K4去甲基化酶的分子机理进行进一步完善。
发表文章:
Qingfeng Chen, Xiangsong Chen, Quan Wang, Faben Zhang, Zhiyong Lou*, Qifa Zhang*, Daoxiu Zhou5*. (2013) Structural basis of a histone H3 lysine 4 demethylase required for stem elongation in rice. PLoS Genet 9(1): e1003239.
