核糖体RNA甲基转移酶RlmG和RsmE的结构和功能研究
核糖体RNA的甲基化修饰在原核生物和真核生物中普遍存在,这一修饰与生物体内蛋白表达调控以及细菌对抗生素的抗药性密切相关。对这一过程的研究将有助于我们深入理解这一过程在调节原核生物和真核生物DNA转录后的修饰和蛋白质合成过程的重要作用,也为进一步开发更有效的抗生素药物提供基础数据。RlmG(m2G1835)和RsmE(m3U1498)是大肠杆菌体内与蛋白表达的调控密切相关的两个核糖体RNA (rRNA)甲基转移酶,中科院高能物理所蛋白质结构研究中心董宇辉研究员领导的团队对这两个蛋白的结构和功能进入了深入的研究,相关的研究成果分别发表于2012年度的《RNA》和《Journal of Molecular Biology》上。
该研究组依托北京同步辐射装置生物大分子实验站站解析了这两个蛋白的高分辨率晶体结构。通过分析RlmG-AdoMet复合物的结构发现,它是由两个同源的N端结构域 (NTD) 和C端结构域 (CTD)串联而成。凝胶迁移电泳实验表明,全长和NTD能有效地结合底物RNA,而CTD不能结合RNA。通过分子模拟方法构建了RlmG-AdoMet-rRNA三元复合物的模型,反应前后底物RNA的构象会发生明显的变化, rRNA会首先解旋成单链构象,随后靶标G1835“滑进”活性位点。rRNA位于两个结构域形成的“凹槽”中间,结合在主要由NTD形成的正电荷区域。该研究揭示了RlmG介导的一种新型催化机制: NTD在RNA的识别和结合过程中起主导作用,而CTD负责结合AdoMet并催化甲基化反应,该反应由两个同源结构域合作完成。
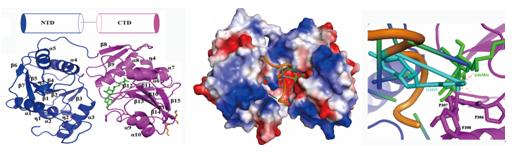
在北京同步辐射装置(BSRF)生物大分子试验站收集RlmG的晶体衍射数据,采用硒代甲硫氨酸标记法解析分辨率2.3 Å的RlmG-AdoMet复合物结构。RlmG由两个高度同源NTD和CTD结构域串联而成。在RlmG-AdoMet-rRNA复合物模型中,rRNA位于两个结构域形成的凹槽中间,结合在主要由NTD形成的正电荷区域。
RsmE是由催化结构域和底物RNA识别结构域这两部分组成,其中在催化结构域中有一个“三叶草”形纽结,组成催化活性中心。采用小角散射方法研究了RsmE在溶液中的构象,与晶体结构的二聚体相比,它的二聚体溶液构象具有灵活性强和亚基间接触面积小的特点,这种柔性构象对底物RNA的识别和结合有重要作用。通过等温滴定量热方法研究RsmE对AdoMet的结合特性发现,两个AdoMet竞争性地结合RsmE二聚体中的两个活性位点,结果只有一个AdoMet能与RsmE结合。在此基础上,构建了RsmE-AdoMet-UMP三元复合物模型,在该模型中AdoMet和UMP结合到二聚体接触面上的三叶草结构附近,采用定点突变方法鉴定出了影响RsmE酶活性的关键氨基酸位点。这项研究揭示了RsmE催化的反应是由它的两个单体密切协作完成的,二聚化对保持它的活性是必需的。
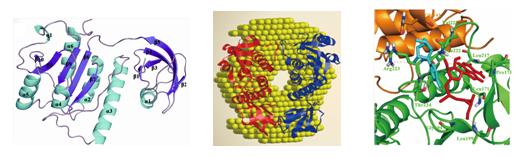
在BSRF生物大分子试验站收集RsmE的晶体衍射数据,采用分子置换法解析分辨率2.25 Å的结构。RsmE是由较大的催化结构域和较小的底物RNA识别结构域这两部分组成。在BSRF小角散射试验站上研究了RsmE的溶液构象,它在溶液中形成灵活性强的二聚体溶液构象,这种柔性构象对底物RNA的识别和结合有重要作用。在RsmE二聚体中,只有一个AdoMet和底物UMP结合到二聚体接触面上的三叶草结构附近。
发表文章:
[1] Zhang H, Gao Z, Wei Y, Wang W, Liu G, Shtykova E, Xu J and Dong Y. (2012) Structural insights into the function of 23S rRNA methyltransferase RlmG (m2G1835) from Escherichia coli. RNA. 18: 1500-1509.
[2] Zhang H, Wan H, Gao Z, Wei Y, Wang W, Liu G, Shtykova E, Xu J and Dong Y. (2012) Insights into the Catalytic Mechanism of 16S rRNA Methyltransferase RsmE (m3U1498) from Crystal and Solution Structures. Journal of Molecular Biology. 423: 576-589.
